- 软件大小:14.2MB
- 软件语言:简体中文
- 软件类型:国产软件
- 软件类别:其它行业
- 更新时间:2018-10-16
- 软件授权:免费版
- 官方网站://www.9553.com
- 运行环境:XP/Win7/Win8/Win10
- 标签:DNAMAN 9 生物新信息软件
DNAMAN 9破解版是一款功能强大的生物综合序列分析软件,该软件能够满足您扩增模板DNA要求的PCR引物,而且从文本文件导入寡核苷酸序列是将大量oligo记录添加到数据库的便捷工具。另外DNAMAN使用序列通道将有效序列保存在计算机内存中。序列通道通过减少磁盘访问时间来提高分析效率。因此小编在这里为大家准备了破解版,教程如下,有需要的赶紧来下载吧!

1、下载软件包解压,双击“DNAMAN_setup.exe”程序安装软件,点击next。

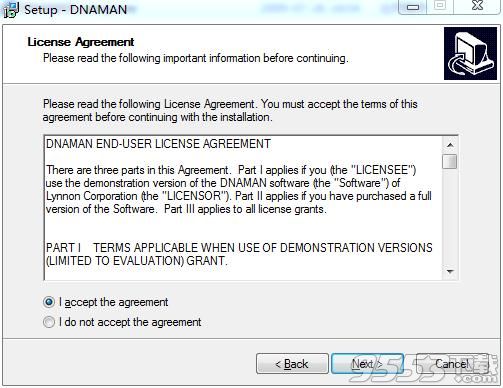
2、选择I accept接受协议,点击next。
3、选择安装位置,默认在C盘建议安装在其他位置。

4、点击install安装软件。

5、正在安装软件,请稍等。
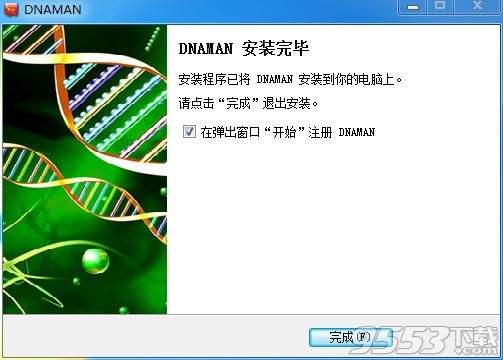
6、安装完成,取消勾选,点击finish。

7、安装完成后,运行“patch.exe”汉化破解补丁,点击下一步。

8、点击完成,将弹出窗口开始注册软件。
9、点击开始注册软件。
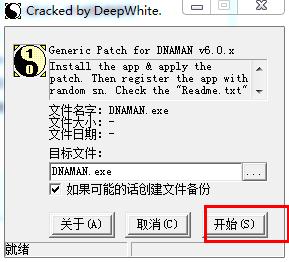
10、汉化补丁注册成功,汉化破解完成。

11、运行桌面快捷方式,如果还是显示需要填写序列号,任意输入数字即可破解。

注意:为避免升级后程序界面变回英文,已禁用程序升级功能;如果您要升级程序,只需将目录下的LBUpdate.dll重命名为LBUpdate.exe 后运行即可。
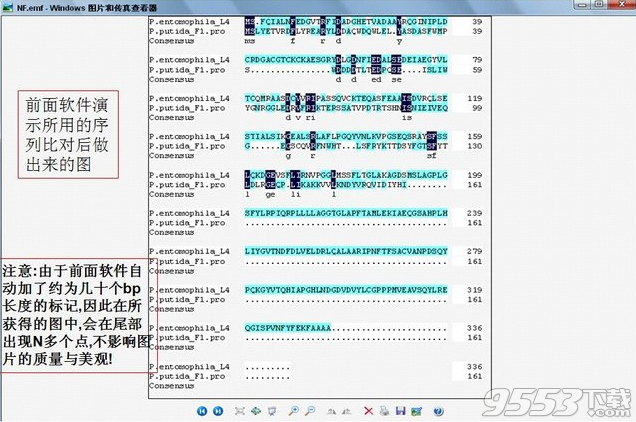
两个引物互补
您可以使用DNAMAN检查两个寡核苷酸序列的互补性。 要执行此功能,第二个引物应通过选择引物|载入DNAMAN存储器 两个Primer互补命令。
DNAMAN搜索两个引物之间的互补序列。 结果表明两个引物之间的连续和不连续的互补序列。
PCR引物设计
您可以使用DNAMAN选择满足您扩增模板DNA要求的PCR引物。 设计了许多参数来筛选PCR引物,例如PCR产物大小,引物长度,Tm,GC组成和盐浓度的范围。 其他参数用于排除“低质量”引物。
导入和导出记录
从文本文件导入记录
从文本文件导入寡核苷酸序列是将大量oligo记录添加到数据库的便捷工具。
DNAMAN将记录的信息列入七个字段:名称,来源,备注,长度,GC含量,熔解温度和序列。如果数据库中有大量记录,则可以使用前四个字段作为排序键,以便在“记录列表”框中选择性地显示记录。
导向不匹配
定向错配可以通过在突变附近的位点引入单次或双重错配来创建或去除当前DNA序列或其变体上的限制性位点。 使用此功能,您可以创建或破坏限制性位点,以区分野生型等位基因与常见突变体等位基因。
1、DNA和蛋白质序列编辑
2、DNA序列转化
3、多序列比对,对齐编辑和分析
4、系统树分析
5、DNA或蛋白质序列的点阵比较
6、DNA序列组装和编辑
7、BLAST通过网络界面在Intranet / Internet Server上进行搜索
8、序列和数据库中的增强型图案搜索
9、SiRNA选择器
10、限制分析
11、绘制序列图与出版品质
12、限制模式预测
13、电子克隆
14、从限制片段重建限制图
15、静音突变分析创建/破坏限制性位点
16、导向不匹配以创建/破坏限制站点
17、翻译和密码子使用分析
18、蛋白质疏水性/亲水性分析
19、蛋白质表征:等电点的序列组成和预测
20、蛋白质二级结构预测
21、反向翻译
22、PCR和测序引物的设计
23、表征DNA或引物序列的热力学性质
24、分歧分析
25、管理寡核苷酸,DNA和蛋白质数据库
26、生成随机序列
序列通道
DNAMAN使用序列通道将有效序列保存在计算机内存中。序列通道通过减少磁盘访问时间来提高分析效率。序列加载到序列通道后,您可以执行许多功能分析,如限制性分析,翻译分析,序列搜索和比较以及PCR引物设计。在DNAMAN中有20个通道,它们中的任何一个都可以被选作默认序列通道。默认顺序是您当前正在处理的顺序。单击序列通道栏上的数字将默认激活相应的通道。
DNA和蛋白质序列都可以加载到通道中。 DNAMAN检查序列的组成以确定其是DNA还是蛋白质片段。 DNA序列自动加载到通道中。蛋白质序列将被用户确认,而DNA序列则不会。如果序列类型未正确定义,您可以使用分析定义功能更改序列类型。信息/序列栏中可以显示默认频道中的序列。
菜单
DNAMAN有九个主菜单显示在屏幕上方。每个主菜单都有几个菜单,其中一些菜单有子菜单。执行功能的命令被描述为主菜单|菜单|子菜单。
例:
a)选择File | Open命令打开一个DNAMAN文件。
b)选择序列|加载顺序|从数据库命令将数据库中的序列记录加载到默认序列通道中。
工具栏
该工具栏设计用于最常用的菜单命令,并允许用户轻松访问它们。您可以使用Info |中的命令显示或隐藏工具栏查看工具栏菜单。
文件
在DNAMAN中有七种文档类型。
1)文本(序列)文件
序列文件是一般文本格式(文本文件)。文本文档显示在文本窗口中。 DNAMAN提供了一个用于序列编辑的标准文本编辑器。序列应保存为纯文本文件,并将“.seq”扩展名添加到文件名中。
2)限制地图文件
限制图用于保存DNA图(*。dmp)。 DNAMAN地图文件是二进制格式并存储限制地图对象。 DMP文件只能用DNAMAN打开和编辑。您也可以将DMP文件作为图形绘制对象复制到任何接受绘图的其他程序。
3)点矩阵文档
DNAMAN将二进制DMF文件中的点阵数据保存为无法作为文本文件打开。
4)多重对齐文件
DNAMAN以MSD(* .msd)格式保存多个序列文档。 MSD文件采用文本格式,并带有用于DNAMAN(MASED)的多重对齐编辑器的特殊关键字。您可以在MASED中加载MSD文件并执行多个序列分析。如有必要,您也可以在文本编辑器中打开MSD文件。
5)序列组装文件
序列组装文档以SAF格式(* .saf)保存。 SAF文件也是文本格式,带有用于DNAMAN序列组装编辑器的特殊关键字。装配编辑器用于编辑和可视化重叠群。如有必要,您也可以在文本编辑器中打开SAF文件。
6)跟踪文件文件
DNAMAN可用于查看和分析自动化DNA测序仪生成的色谱图文件,例如ABI系列PE BioSystem。 DNAMAN可以打开ABI和SCF文件,也可以将编辑好的序列保存到这些文件中。
7)系统发育树文件
DNAMAN以扩展名PTR保存文本格式的系统发育树文件。系统发育树文件可以作为文本打开或编辑。
状态栏
状态栏显示在DNAMAN窗口的底部。 要显示或隐藏状态栏,请使用视图菜单中的状态栏命令。
状态栏的左侧区域描述了使用箭头键浏览菜单时菜单项的操作。 此区域同样显示描述工具栏按钮按下时的动作的消息,然后释放它们。 如果在查看工具栏按钮命令的描述后,希望不执行该命令,则在指针离开工具栏按钮时释放鼠标按钮。
状态栏的右侧区域表示下列哪个键被锁定:
指标描述
CAP Caps Lock键锁定。
NUM Num Lock键被锁定。
SCRL Scroll Lock键锁定。
工具栏
工具栏显示在应用程序窗口的顶部,菜单栏下方。 该工具栏提供了对DNAMAN中使用的许多工具的快速鼠标访问,
要隐藏或显示工具栏,请单击视图菜单中的工具栏。
开始使用DNAMAN
了解DNAMAN
序列编辑
DNAMAN提供了一个文本编辑器来编辑序列文件。您可以添加或删除文件中的任何文本或序列。您也可以使用复制和粘贴功能来编辑文件。一些序列分析结果也显示为文本文件,因此可以使用编辑器编辑这些结果。当你打开一个序列文件时,序列可以直接加载到当前通道。如果自动加载选项关闭,分析功能不可立即使用。在分析之前,您必须手动将序列加载到通道中。
序列分析
您可以选择一个序列并对其进行多种分析。该序列必须加载到序列通道中。 DNAMAN提供了20个序列通道来保存内存中的活动序列。您可以同时分析和比较通道中的所有序列。您可以在分析过程中将一个序列切换到另一个序列,而不必回到磁盘文件。
序列比对和比较
您可以直接使用磁盘文件进行多重对齐和序列组装。没有必要将这些序列加载到通道。要执行这些任务,请在菜单栏上选择相应的命令,选择序列文件并填写适当的参数。 DNAMAN会记住这些分析中使用的文件。您可以重新分析这些序列而无需返回到磁盘文件。
快速启动DNAMAN
如果您第一次使用DNAMAN,您可以按照下面的说明进行操作。
要执行序列分析:
1)启动DNAMAN。序列文件示例会自动打开,您可以转到步骤3.否则,请选择Sequence |加载顺序|从序列文件菜单中。
2)从打开文件对话框中选择Example1.Seq文件。单击打开将Example1加载到序列通道1。
3)选择限制|限制分析菜单对Example1执行限制分析。
4)限制性分析后,选择蛋白质|翻译菜单将Example1翻译成氨基酸序列。
要绘制没有序列的限制图:
选择限制|绘制地图菜单。质粒图谱在限制图窗口中绘制。双击窗口以显示地图属性并进行修改。
提示:限制地图中的所有对象都可以通过拖放来移动。限制网站和文本对象在被选中时可以对齐。
将待分析的序列装入Channel,点击要分析的Channel,然后通过Restriction/Analysis 命令打开对话框.
参数说明如下:
Results 分析结果显示
其中包括:
Show summary(显示概要) Show sites on sequence(在结果中显示酶切位点)
Draw restriction map(显示限制性酶切图)Draw restriction pattern(显示限制性酶切模式图)
Ignore enzymes with more than(忽略大于某设定值的酶切位点)
Ignore enzymes with less than(忽略小于某设定值的酶切位点)
Target DNA (目标DNA 特性)
circular(环型DNA),dam/dcm methylation(dam/dcm 甲基化)
参数说明如下:
Enzyme
代表(enzyme data file),点击旁边的下拉按钮,出现两个默认选项,restrict.enz 和dnamane.enz,
如果添加过自制的酶列表,则附加显示自制酶列表文件名.其中restrict.enz 数据文件包含180 种
限制酶,dnamane.enz 数据文件包含2524 种限制酶.选择其中一个数据文件,相应的酶在左边的 显示框中列出(按酶名称字母表顺序),鼠标双击酶名称,则对应的酶被选中,在右边空白框中列 出.要自制酶切列表,可以从左边酶列表中双击鼠标选择多种酶(例如puc18 multiple cloning sites),然后点击按钮出现下列对话框:输入要保存酶列表的文件名,点击按钮即可保存.自制酶列表可以方便分析特定的酶切位点.
Cutter 酶切识别序列长度;End 酶切产生的末端,其中包括,Blunt(平头末端),5’Overhang
(5’突出粘性末端),3’Overhang(3’突出粘性末端),系统根据cutter 和end 的设定情况,在左边酶列表 中显示符合条件的酶.最后,点击按钮执行操作.

图1

图2
图3
图4
图5

图6

图7

图8

图9

图10